Новый «полный» геном шимпанзе показывает разницу в 14,9% с человеческим геномом
Новаторская статья в журнале Nature сообщает о «Полном секвенировании геномов человекообразных обезьян», включая геномы шимпанзе, бонобо, горилл, борнейских орангутанов, суматранских орангутанов и сиамангов. Я упомянул об этом в своей статье вчера, отметив, что эволюционный символ — знаменитое «1% различие» между человеческим и шимпанзным геномами, широко рекламируемое в научно-популярной литературе и преподавании — рухнул. Исследователи, по каким-то причинам — я не телепат — решили скрыть это поразительное открытие в техническом жаргоне раздела «Дополнительные данные». Теперь перейдем к научным деталям.
Вы можете подумать: «Разве эти геномы не были секвенированы давно?» Ответ — и да, и нет. Да, мы уже секвенировали геномы этих видов в прошлом, но, как объясняется в статье, «из-за повторяющегося характера геномов человекообразных обезьян полные сборки не были достигнуты. Текущие референсы не имеют разрешения последовательностей в некоторых из самых динамичных регионов генома, включая области, соответствующие видоспецифичным семействам генов».
Или, как говорится в сопровождающей пояснительной статье:
«Раньше ученые расшифровывали сегменты геномов нечеловекообразных обезьян, но им никогда не удавалось собрать полную последовательность для какого-либо вида. Однако в текущем исследовании [Катерина] Макова и ее коллеги использовали передовые методы секвенирования и алгоритмы, которые позволили им прочитать длинные сегменты ДНК и собрать их в последовательность, простирающуюся от одного конца хромосомы до другого, без пробелов. "Этого никогда не делали раньше", говорит Макова».
Другими словами, полные геномы человекообразных обезьян никогда не были полностью секвенированы. И они использовали человеческий геном в качестве референсной последовательности, что делало геномы обезьян более похожими на человеческий, чем они есть на самом деле.
Вы мне не верите?
Из технической статьи:
«Большинство предыдущих сравнительных исследований геномов человекообразных обезьян были ограничены сопоставлением некачественных сборок с более качественным человеческим геномом. В результате были внесены систематические ошибки, связанные с человеческим референсом».
Это согласуется с тем, что Национальный центр биотехнологической информации (NCBI) заявил в 2007 году о раннем черновике генома шимпанзе:
«Контиги были собраны с использованием человеческого генома в качестве ориентира и поэтому "очеловечены" в своей структуре. Это важное различие, так как некоторые последовательности, такие как вставки, делеции и дупликации генов, могут быть неточно представлены в текущей сборке шимпанзе».
Таким образом, до сих пор все версии геномов шимпанзе и других человекообразных обезьян были фактически «очеловечены», потому что они «собирались с использованием человеческого генома в качестве ориентира». Это заставляет их казаться более похожими на человеческий геном, чем они есть на самом деле. Могут ли эти новые черновики геномов обезьян помочь исправить проблему?
Проблема решена?
Другая пояснительная статья в Nature, похоже, предполагает, что эти «полные» черновики геномов обезьян докажут, что они менее похожи на человеческий геном, чем утверждалось:
«Вскоре после завершения первого секвенирования человеческого генома в 2003 году была выпущена сборка шимпанзе. За ней последовали сборки других человекообразных обезьян, таких как горилла, суматранский орангутан и бонобо, а также малых обезьян, менее родственных человеку, чем человекообразные. Эти геномы предоставили ценную возможность каталогизировать генетические различия, накопленные в ходе эволюции обезьян, включая изменения, уникальные для человека. Но поскольку эти первоначальные релизы были неполными черновиками, сравнения можно было проводить только между правильно разрешенными участками генома. Эти исследования поэтому фокусировались только на относительно небольших различиях и исключали чрезвычайно повторяющиеся последовательности и крупномасштабные структурные различия, такие как инверсии и дупликации геномных последовательностей».
Последнее предложение, похоже, намекает, что предыдущие сравнения человеческих и обезьяньих геномов «фокусировались только на относительно небольших различиях» и «исключали» участки, содержащие «крупномасштабные структурные различия». Таким образом, можно ожидать, что эти новые «полные» геномы обезьян выявят гораздо большие различия по сравнению с человеческим геномом.
Новые геномы обезьян и человеческий геном
При чтении технической статьи прямое сравнение геномов обезьян и человека найти сложно. Этот отрывок, кажется, ближе всего к нему:
«В целом, сравнения последовательностей между полными геномами человекообразных обезьян выявили большую дивергенцию, чем предполагалось ранее (Дополнительные заметки III-IV). Действительно, 12,5–27,3% генома обезьяны не выравнивались или были несовместимы с простым однозначным сопоставлением, что привело к появлению пробелов».
Что именно это означает? Во-первых, они признают, что «сравнения последовательностей между полными геномами человекообразных обезьян выявили большую дивергенцию, чем предполагалось ранее». Но техническая статья в Nature считает людей «человекообразными обезьянами», поэтому подразумевается, что сравнение «геномов обезьян» включает сравнения между человеческими и обезьяньими (т.е. нечеловеческими гоминоидами) геномами. Поэтому в дальнейшем я буду называть людей «людьми», а нечеловеческих гоминоидов — «обезьянами», как это делают большинство обычных людей.
Интересно, что две препринтные версии статьи (v1 и v2), опубликованные в прошлом году на BioRxiv (которые, предположительно, являются первоначальной и исправленной версиями рукописи, отправленной в Nature), предваряют этот результат двумя предложениями:
«Часто цитируемая статистика о ~99% идентичности последовательностей между шимпанзе и человеком верна для большей части генома, если рассматривать однонуклеотидные варианты (SNV). Однако сравнения T2T-геномов предполагают гораздо более сложную оценку».
T2T означает исследование «от теломеры до теломеры» — т.е. исследование всей хромосомы по всему геному. Эти предложения, очевидно, были удалены во время редактуры для публикации в Nature — интересное редакторское решение. Так что же статья говорит о различии между людьми и шимпанзе?
Как мы увидим, приведенное выше утверждение — что «сравнения последовательностей между полными геномами человекообразных обезьян выявили большую дивергенцию, чем предполагалось ранее» — верно. Но оно не раскрывает масштаба различий между человеческими и обезьяньими геномами, обнаруженных в этом исследовании. Поэтому перейду к сути:
Взгляните на эти цифры: «12,5–27,3%». Те же цифры снова появляются глубоко в Дополнительных данных, где сравниваются различные геномы обезьян с человеческим. Их можно найти, если знать, где искать, но стоит ли говорить «закопаны» — или «скрыты»? Насколько я могу судить, Дополнительные данные сообщают, что геном обезьяны, наиболее похожий на человеческий, — это геном шимпанзе. И он показывает «разрыв-дивергенцию» в 12,5% — т.е. различие — с человеческим геномом! А если посмотреть на «разрыв-дивергенцию», где человеческий геном является целью, а шимпанзе — запросом, различие составляет 13,3%. Позвольте мне прояснить: согласно этому исследованию, человеческий и шимпанзный геномы не совпадают на 98,8% (или различаются на 1,2%), как, например, утверждает Национальный музей естественной истории Смитсоновского института (см. мой «Путеводитель для посетителей»). На самом деле они схожи не более чем на 87,5% — т.е. человеческий и шимпанзный геномы различаются как минимум на 12,5%, если не на 13,3%! Фактически, различие в 13,3% более актуально, поскольку отражает, насколько вся человеческая сборка похожа на геном шимпанзе.
Что именно такое «разрыв-дивергенция»?
Прежде чем двигаться дальше, я хочу разобраться, что именно авторы подразумевают под «разрыв-дивергенцией» или «разрыв-различием». В статье «разрыв-дивергенция» определяется следующим образом:
«Разрыв-дивергенция определяется как доля позиций в целевой гаплотипе, которые не выравниваются с другим гаплотипом, что может быть связано с биологическими процессами (например, потерей/приобретением генов и вставками/делециями), отсутствующими данными или техническими проблемами (например, ошибками выравнивания из-за структурных вариаций, повторяющихся элементов и т.д.)».
Так как они определяют разрыв-дивергенцию? Насколько я могу судить, она основана на разделении целевого генома в выравнивании генома на сегменты по 1 миллиону пар оснований (1 Мпн) и подсчете того, сколько оснований в каждом сегменте 1 Мпн не имеют соответствующего основания в запрашиваемом геноме, который был выровнен с ним. Если весь сегмент 1 Мпн не имеет выравнивания с целевым геномом, его разрыв-дивергенция составляет 100%. Если 10 000 п.н. не выровнены, разрыв-дивергенция составляет 1%; если 1 000 п.н. не выровнены — 0,1% и т.д. Согласно результатам исследования, средняя разрыв-дивергенция в каждом сегменте 1 Мпн человеческого генома (как цели), выровненного с геномом шимпанзе (как запроса), составляет 12,5%. Таким образом, 12,5% оснований в человеческом геноме не имеют соответствующих оснований в геноме шимпанзе в рамках полного выравнивания генома.
Приведенная ниже иллюстрация (созданная для наглядности, а не из исследования) помогает показать различия между «SNV» и «разрывами» между двумя геномами:
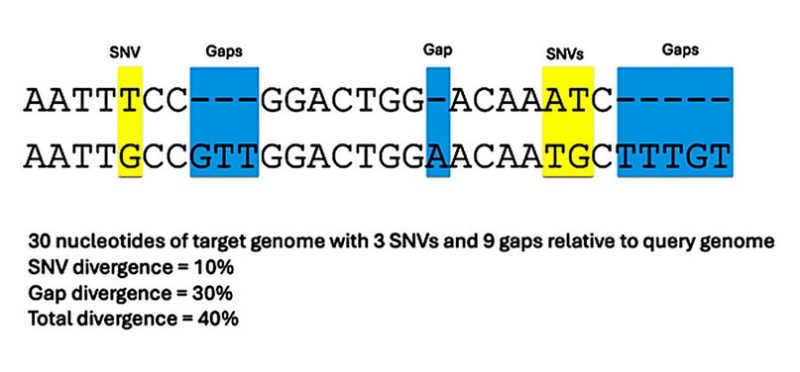
Как видно, «разрывы» представляют нуклеотиды или сегменты нуклеотидов, которых просто нет в одном геноме или другом, тогда как «SNV» представляют нуклеотиды, которые существуют, но различаются. Эти два типа различий можно суммировать, чтобы вычислить общее различие между геномами.
Верхняя оценка
И почему в основном тексте указан диапазон от 12,5% до 27,3%? Потому что верхняя оценка невыравниваемости между геномом гориллы и человеческим геномом составляет колоссальные 27,3%. Фактически, если мы посмотрим на Дополнительный рисунок III.12, мы найдем следующие проценты «разрыв-дивергенции» между различными геномами обезьян при сравнении с человеческим геномом (неполовые хромосомы):
- Суматранский орангутан (Pongo abelii) vs человек: 15,4% и 16,5% «разрыв-дивергенции» (т.е. минимальное различие)
- Горилла (Gorilla gorilla) vs человек: 17,9% и 27,3% «разрыв-дивергенции» (т.е. минимальное различие)
- Бонобо (Pan paniscus) vs человек: 12,5% и 14,4% «разрыв-дивергенции» (т.е. минимальное различие)
- Шимпанзе (Pan troglodytes) vs человек: 12,5% и 13,3% «разрыв-дивергенции» (т.е. минимальное различие)
Видите, как легко обобщить эти данные? Это огромные открытия для широкой публики, но техническая статья в Nature и две пояснительные статьи в Nature не смогли четко выделить эти моменты. Они скрыли их в техническом жаргоне и отсутствии ясности глубоко в Дополнительных данных, а предложение о «часто цитируемой статистике ~99% идентичности последовательностей» было удалено во время редактуры статьи. Nature, я уверен, не является журналом, который редактируется хаотично. Это были осознанные решения кого-то в процессе редактирования. Отсутствие ясности просто невероятно.
Технические детали
Глубоко в Дополнительных данных мы находим Рисунок III.12, который объясняет разрыв-дивергенцию между разными видами.
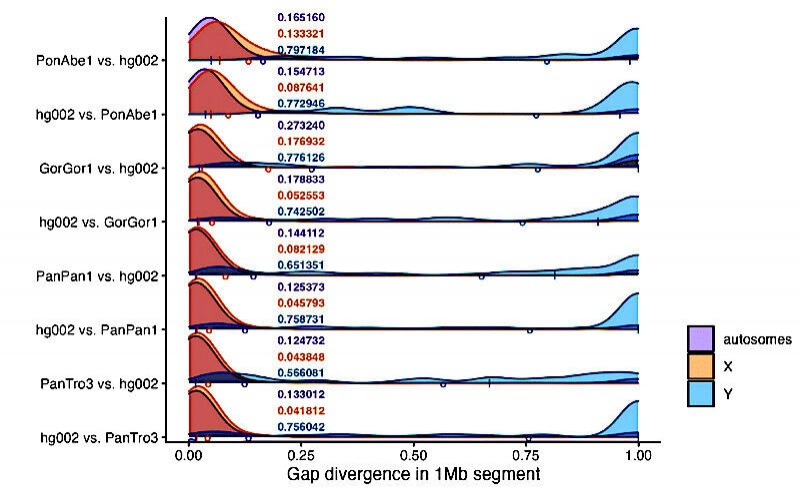
Подпись гласит: «Графики показывают сегменты 1 Мпн, сгруппированные по разрыв-дивергенции для каждого попарного выравнивания», где попарное выравнивание — это попытка сопоставить две последовательности для определения степени их сходства или различия. Таким образом, мы видим прямое измерение минимальной степени различия между двумя геномами.
Добавление однонуклеотидных вариаций (SNV)
Но есть еще один тип вариаций между геномами, также идентифицированный в статье — однонуклеотидные различия (называемые «однонуклеотидными вариациями» или иногда «короткими нуклеотидными вариациями», SNV). Опять же, в Дополнительных данных мы находим Рисунок III.11, который показывает процент SNV между человеческим и различными геномами обезьян, о которых сообщается в этом исследовании. Вот что они обнаружили:
- Суматранский орангутан (Pongo abelii) vs человек: ~3,6% различий
- Горилла (Gorilla gorilla) vs человек: 1,9–2,0% различий
- Бонобо (Pan paniscus) vs человек: 1,5–1,6% различий
- Шимпанзе (Pan troglodytes) vs человек: 1,5–1,6% различий
Если мы добавим разрыв-дивергенцию к различиям SNV, мы получим следующие общие степени различия между человеческим и обезьяньими геномами:
- Суматранский орангутан (Pongo abelii) vs человек: ~19–20,1% различий
- Горилла (Gorilla gorilla) vs человек: ~19,8–29,3% различий
- Бонобо (Pan paniscus) vs человек: ~14,0–16,0% различий
- Шимпанзе (Pan troglodytes) (цель) vs человек: ~14,0% различий
- Человек (цель) vs шимпанзе (Pan troglodytes): ~14,9% различий
Теперь мы видим, что общее различие между человеческим геномом и геномом шимпанзе составляет около 14,9%. Это представляет гораздо большую степень различия, чем часто утверждаемая статистика о том, что мы отличаемся от шимпанзе всего на 1%!
Это окончательный результат?
Несомненно, требуется больше анализа, чтобы определить, в какой степени нуклеотиды демонстрируют «точные однозначные совпадения» между человеческим и шимпанзным геномами даже в регионах, которые можно было легче выровнять. Поэтому я подозреваю, что степень различия между человеческим и шимпанзным геномами может увеличиться в будущем.
Пока же мы можем с уверенностью сказать, что это последнее исследование показывает, что человеческий и шимпанзный геномы различаются как минимум на 14,9%. Это означает, что человеческий и шимпанзный геномы как минимум на порядок более различны, чем обычно утверждается.
Конечно, мы говорим здесь о 44 неполовых хромосомах в человеческом геноме. Также стоит отметить, что по сравнению с шимпанзе человеческая Y-хромосома имеет колоссальную разрыв-дивергенцию в 56,6% (и 3,9% различий SNV), а человеческая X-хромосома имеет разрыв-дивергенцию в 4,4% (и 1,1% различий SNV). Но и это все скрыто в Дополнительных данных.
Все это — революционные открытия, и очень жаль, что Nature не сообщил о данных четко и сделал все это так труднонаходимым, используя жаргон, который большинство неспециалистов не поймет. Почему они так поступили? Важно понимать, что публикация научных статей иногда напоминает изготовление колбасы: процесс часто грязный, и окончательная форма, которую вы читаете, обычно представляет собой компромиссный язык, с которым согласились все авторы, рецензенты и редакторы — и он может не отражать точку зрения каждого автора статьи. Поэтому, возможно, некоторые авторы этого исследования предпочли бы изложить выводы более прямо. Но мы все равно можем спросить: Почему Nature не изложил результаты ясно и не позволил фактам говорить самим за себя?
Я подозреваю, что это радикальное открытие имеет последствия — для человеческой исключительности, для надежности широко рекламируемых утверждений и многого другого — которые будут обсуждаться еще долго. И, возможно, для некоторых в мире науки и научной журналистики, особенно для тех, кто продвигал теперь опровергнутую цифру о всего 1% различия с шимпанзе, эти обсуждения могут быть нежелательными.
Кейси Ласкин
EN Evolution News

Comments are closed.